Impfstoffe in Rekordzeit: Künstliche Intelligenz leistet Beitrag zur Wissenschaft
Die schnelle Entwicklung von zwei hochwirksamen Covid-19-Impfstoffen war dank Künstlicher Intelligenz und innovativer Zusammenarbeit zwischen Forschenden auf der ganzen Welt möglich. Darunter auch solche aus der Schweiz.
Künstliche Intelligenz (KI) stellt uns vor ethische Herausforderungen. Aber sie könnte auch die Wissenschaft revolutionieren und einige der komplexesten Probleme der modernen Biologie lösen.
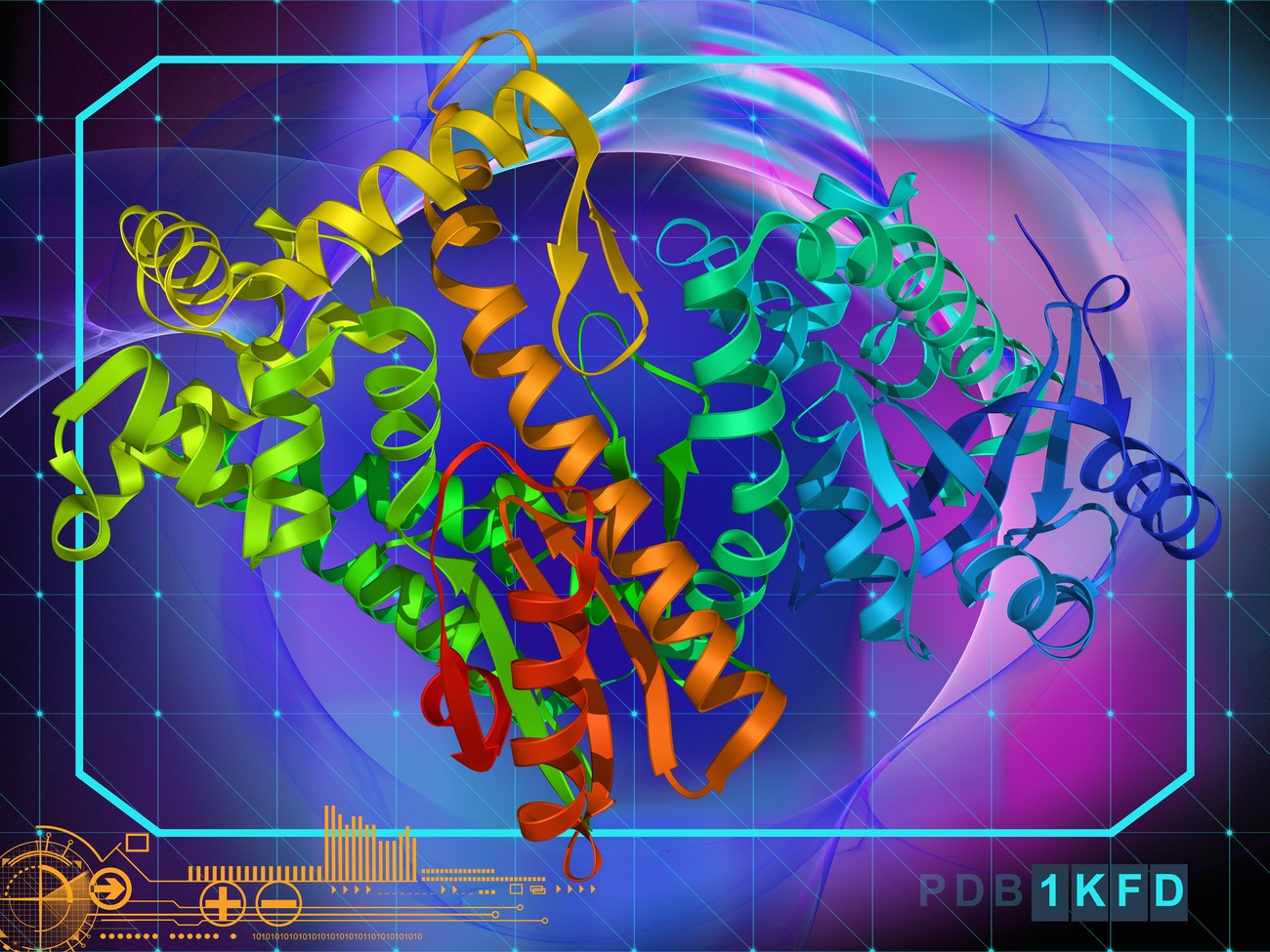
An erster Stelle: Die Vorhersage der Struktur unbekannter Proteine, um die Geheimnisse der Zellen und der Krankheiten, die sie betreffen, zu entschlüsseln. In jüngster Zeit haben Proteine wegen ihrer zentralen Rolle bei der Entwicklung neuer Impfstoffe gegen Covid-19 von sich reden gemacht.
Die Form von Proteinen experimentell zu bestimmen, ist ein langwieriger und mühsamer Prozess, der monatelange Forschung und einen hohen Aufwand erfordert. Diese Informationen sind allerdings wichtig, um neue Viren zu untersuchen, ihr Verhalten zu verstehen und wirksame Impfstoffe zu entwickeln. Die Fähigkeit der Wissenschaftlerinnen und Wissenschaftler, Proteinstrukturen mit Hilfe von Berechnungsmethoden vorherzusagen, hat diesen Prozess nicht nur schneller, sondern auch genauer gemacht.
Dank der jüngsten Fortschritte in der Künstlichen Intelligenz ist es möglich, die dreidimensionalen Strukturen hochkomplexer Zielproteine mit grosser Genauigkeit vorherzusagen. Ein grosser Meilenstein wurde erreicht, als AlphaFold2, das KI-System des Londoner Unternehmens DeepMind (seit 2014 im Besitz von Google), es ermöglichte, in kürzester Zeit mehrere Proteinstrukturen von SARS-CoV-2 zu bestimmen, einem Virus, über das bis vor wenigen Monaten nur sehr wenig bekannt war.
Wendepunkt für die Wissenschaft
Die unermüdliche Arbeit der Forschenden und die Zusammenarbeit auf internationaler Ebene – mit Hilfe modernster KI-Technologien wie der von DeepMind – ermöglichten es, schnell auf die Pandemie zu reagieren. Derzeit befinden sich bis zu 60 potenzielle Impfstoffe in der klinischen Entwicklung. Laut WeltgesundheitsorganisationExterner Link wurden bisher drei Impfstoffe von nationalen Zulassungsbehörden für den grossflächigen Einsatz zugelassen.
Biomedizinerinnen und -mediziner betrachten diesen Moment als einen Wendepunkt für die Wissenschaft. «Das ist ein unglaubliches Resultat», kommentiert Torsten Schwede, Vizepräsident für Forschung an der Universität Basel und Leiter der Forschungsgruppe am Schweizerischen Institut für Bioinformatik SIB.
Das SIB hat das SWISS-MODEL entwickelt, einen vollautomatischen Server zur Modellierung von Proteinstrukturen, der von Forschenden weltweit genutzt wird. Die Erfolge von DeepMind wurden auch durch die Fortschritte des letzten Jahrzehnts auf dem Gebiet der computergestützten Strukturbiologie ermöglicht, bei der das Schweizer System SWISS-MODEL ein Pionier war.
«Der offene Austausch von Informationen innerhalb der wissenschaftlichen Gemeinschaft ermöglichte die Entwicklung von Impfstoffen in Rekordzeit.»
Torsten Schwede
Menschen und Software
Warum rücken Proteine in den Mittelpunkt des medizinischen und wissenschaftlichen Interesses? Sie sind klein, aber von grundlegender Bedeutung, denn Proteine untermauern die chemischen und biologischen Prozesse der menschlichen Zellen und jedes lebenden Organismus. Die Aminosäuren, aus denen Proteine bestehen, verbinden sich zu einem spontanen »Origami», das ihre besondere dreidimensionale Struktur bestimmt.
Zu wissen, welche Form Proteine haben, erleichtert die biomedizinische Forschung erheblich, beispielsweise bei der Erforschung menschlicher Krankheiten. Aus diesem Grund werden die von DeepMind erzielten Ergebnisse von der wissenschaftlichen Gemeinschaft als revolutionär angesehen. Es besteht die Hoffnung, dass die Erkenntnisse in die Entwicklung neuer Medikamente und fortschrittlicher pharmakologischer Behandlungen einfliessen.
Dank SWISS-MODEL waren Forschende weltweit erstmals in der Lage, die dreidimensionale Struktur von noch nicht experimentell erforschten Proteinen selbstständig zu modellieren. 1993 lancierte Manuel Peitsch, Bioinformatiker und Gründer von SWISS-MODEL, eine Idee: Mit Hilfe von Computersimulationssystemen, die kein menschliches Eingreifen erfordern, Strukturinformationen über Proteine zu erhalten und molekulare Funktionen besser zu verstehen.
Zu dieser Zeit erschien dieses Projekt wie Science-Fiction. Heute sind die Softwares dank immer ausgeklügelter Methoden den menschlichen Fähigkeiten in Bezug auf Genauigkeit und Leistung überlegen und werden weltweit eingesetzt. Sie können bereits bekannte Proteinstrukturen mit unbekannten Proteinsequenzen vergleichen, man nennt das «Homologiemodellierung». Jedes Jahr bearbeitet SWISS-MODEL ohne menschliche Aufsicht mehr als eine Million Anfragen für Proteinmodelle.
Proteine sind flexible Objekte. Bewegungen sind für sie oft notwendig, um zu funktionieren. Um ein 3D-Modell eines Proteins mit einer experimentellen Referenzstruktur zu vergleichen, muss das Modell »gedreht» werden, bis eine optimale Überlappung vorliegt.
Bei flexiblen, sich verändernden Objekten ist diese Überlappung jedoch nur schwer zu erreichen. Um dieses Problem zu lösen, entwickelte das SWISS-MODEL-Team einen Wert namens lDDT (Local Distance Difference Test), der den Grad der Übereinstimmung einer Vorhersage mit der Referenzstruktur unabhängig von intramolekularen Bewegungen bewertet. Solche Daten, die ohne menschliche Aufsicht verwendet werden können, sind für die Entwicklung von autonomen Proteinmodellierungsmethoden unerlässlich.
Realität und Science Fiction
Seit drei Jahrzehnten versuchen Forschende, die charakteristische dreidimensionale Form von Proteinen aus ihren Aminosäuresequenzen abzuleiten. Dabei macht die Kenntnis der experimentellen Strukturen von verwandten Proteinen die Modellierung relativ einfach und genau.
In schwierigen Fällen bedeutet jedoch das Fehlen struktureller Informationen über eine bestimmte Proteinfamilie, dass eine sehr komplizierte und oft ungenaue Vorhersage von Grund auf gemacht werden muss. Aber dank AlphaFold2 besteht dieses Problem nicht mehr. »Wir haben gesehen, dass die von DeepMind entwickelte AlphaFold2-Methode sowohl für einfache als auch für sehr komplexe Fälle funktioniert. Das ist ein echter Durchbruch, denn jetzt kann die KI etwas leisten, was zuvor kein Mensch mit fundierten Kenntnissen in der Proteinmodellierung geschafft hat», sagt Schwede.
Das KI-System von DeepMind verwendet fortschrittliche Techniken des maschinellen Lernens, so genannte tiefe neuronale Netze, um die Strukturen von Proteinen direkt aus ihren genetischen Sequenzen vorherzusagen. Dazu lernte das KI-System die Sequenzen und Strukturen von etwa 100’000 bekannten Proteinen, indem es auf experimentelle Daten zurückgriff.
Jetzt ist es in der Lage, sehr genaue Vorhersagen von 3D-Modellen beliebiger Proteine zu machen. Die aussergewöhnlichen Ergebnisse von DeepMind wurden von den Organisatoren des Experiments zu CASP-Proteinstrukturen bestätigtExterner Link (siehe Box), welche die Berechnungs- und Vorhersagekapazität von AlphaFold2 als »beispiellos» bezeichnet haben.
Das CASP (Critical Assessment of protein Structure Prediction) ist ein Experiment, das alle zwei Jahre stattfindet und die Entwicklungen auf dem Gebiet der Proteinstrukturvorhersage international bewertet. Während des kürzlich durchgeführten Experiments im Jahr 2020 (CASP14) wurde die Genauigkeit der Vorhersagemethoden an fast hundert Protein-Targets evaluiert. Die AlphaFold2-Vorhersagen erwiesen sich auch in schwierigen Fällen als sehr genau, wie beispielsweise bei einem bisher unbekannten SARS-CoV-2-Protein, ORF8. SARS-CoV-2 setzt sich aus etwa 30 verschiedenen Proteinen zusammen, von denen über ein Dutzend noch wenig bekannt ist.
Die goldene Regel des Fortschritts
Die starke Unterstützung durch die wissenschaftliche Gemeinschaft und der offene Informationsaustausch im Bereich der computergestützten Strukturbiologie, zu dem auch SWISS-MODEL beigetragen hat, sind einige der Zutaten, die den Erfolg von DeepMind erklären. Laut Torsten Schwede hat das offene Teilen von Berechnungsmethoden und Strukturdaten DeepMind die notwendigen Informationen gegeben, um eines der schwierigsten Probleme in der Bioinformatik zu lösen.
Der Austausch intensivierte sich während der Pandemie und zeigte, wie wichtig die Zusammenarbeit ist, um in deutlich kürzerer Zeit sinnvolle Ergebnisse zu erzielen. »Wir haben während dieser Pandemie viel gelernt. Der offene Austausch von Informationen über SARS-CoV-2 innerhalb der wissenschaftlichen Gemeinschaft ermöglichte die Entwicklung von Impfstoffen in Rekordzeit», so Schwede. Datensilos seien für die Wissenschaft kontraproduktiv.
Sibilla Bondolfi

In Übereinstimmung mit den JTI-Standards













Einen Überblick über die laufenden Debatten mit unseren Journalisten finden Sie hier. Machen Sie mit!
Wenn Sie eine Debatte über ein in diesem Artikel angesprochenes Thema beginnen oder sachliche Fehler melden möchten, senden Sie uns bitte eine E-Mail an german@swissinfo.ch